In die proses om genetiese studies uit te voer, kom ons dikwels onvoldoende RNA-monsters teë, byvoorbeeld vir die bestudering van klein anatomiese orale gewasse, selfs enkelselmonsters, en monsters van spesifieke geenmutasies wat teen baie lae vlakke in menslike selle getranskribeer word.Natuurlik, vir die COVID-19-toets, as die deppers nie op die regte plek of nie genoeg kere tydens steekproefneming is nie, sal die steekproefgrootte baie laag wees, en daarom het die Kommissie van Gesondheid en Gesinsbeplanning twee dae gelede uitgekom en die toets geslaag het, en as die nukleïensuurmonsternemer nie ses monsters geneem het nie, kan jy dit rapporteer.
Die sensitiwiteit van die reagens is belangrik omdat ons hierdie probleem of daardie probleem het, so wat kan ons doen om die sensitiwiteit van RT-PCR te verbeter?
Voordat ons moontlike oplossings bespreek, kom ons noem twee groot komplikasies met die situasie wat ons sopas genoem het.
Eerstens is ons bekommerd oor RNA-verlies wanneer ons slegs 'n paar selpopulasies in ons steekproef het.Indien tradisionele skeidings- en skoonmaakmetodes gebruik word, soos kolommetode of nukleïensuurpresipitasiemetode, is daar 'n groot moontlikheid dat die paar monsters verlore gaan.Een oplossing is om 'n draermolekule, soos tRNA, by te voeg, maar selfs dan is daar geen waarborg dat ons herstel-eksperiment OK is nie.
So wat is 'n beter manier?'n Goeie opsie vir gekweekte selle of mikroanatomiese monsters is om direkte lise te gebruik.
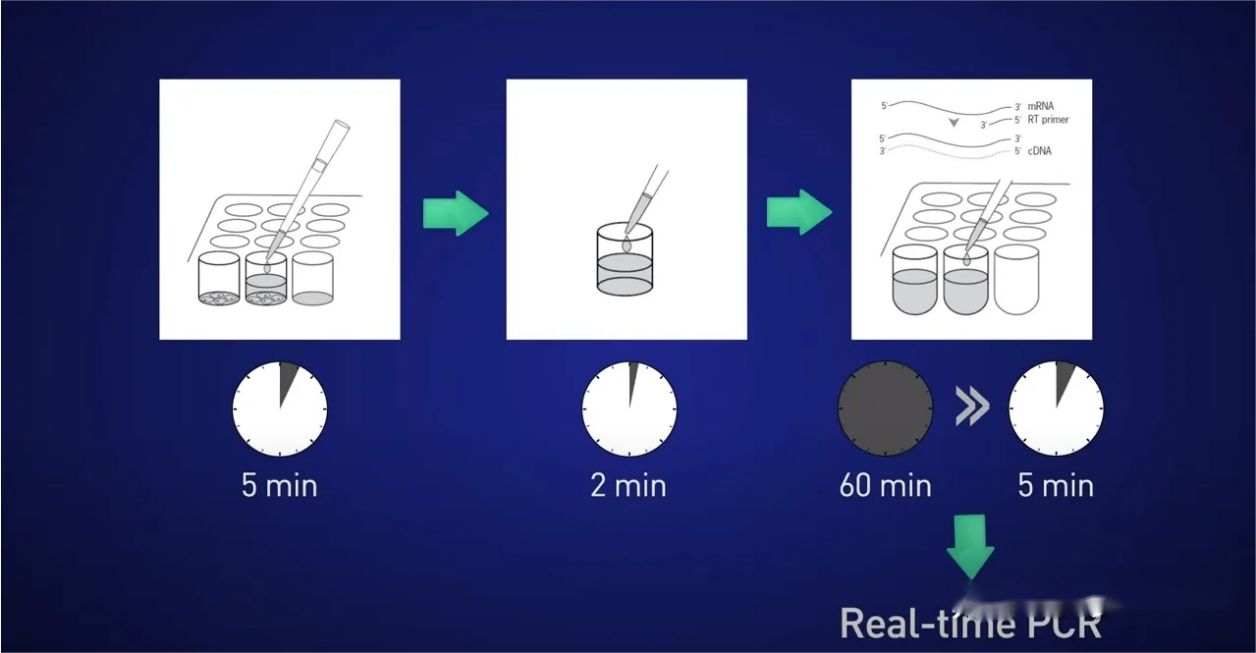
Die idee is om die selle vir 5 minute te verdeel, die RNA in die oplossing vry te stel, dan die reaksie vir 2 minute te stop, dan die lysaat direk by die omgekeerde transkripsie-reaksie te voeg sodat geen RNA verlore gaan nie, en uiteindelik die resulterende cDNA direk te plaas in die intydse reaksie.
Maar wat as ons, as gevolg van 'n beperkte beginpunt of 'n klein hoeveelheid teikengeenuitdrukking, al die RNA kan herwin en steeds nie genoeg sjablone verskaf om 'n goeie intydse sein te kry nie?
In hierdie geval kan die pre-amplifikasie stap baie nuttig wees.
Die volgende is 'n skema om sensitiwiteit na omgekeerde transkripsie te verhoog.Voordat ons begin, moet ons stroomaf vra in watter teikens ons belangstel, om spesifieke primers vir hierdie teikens vir pre-amplifikasie te ontwerp.
Dit kan bereik word deur 'n gemengde onderlaag te skep met tot 100 pare onderlaag en 'n reaksiesiklus van 10 tot 14 keer.Daarom is 'n Master Mix spesifiek ontwerp vir hierdie vereiste nodig om die cDNA wat verkry is vooraf te versterk.
Die rede waarom die aantal siklusse tussen 10 en 14 gestel word, is dat hierdie beperkte aantal siklusse ewekansigheid tussen die verskillende teikens verseker, wat deurslaggewend is vir navorsers wat kwantitatiewe molekulêre inligting benodig.
Na pre-amplifikasie kan ons 'n groot hoeveelheid cDNA kry, sodat die opsporingsensitiwiteit aan die agterkant aansienlik verbeter word, en ons kan selfs die monster verdun en verskeie intydse PCR-reaksies uitvoer om moontlike ewekansige foute uit te skakel.
Postyd: 11-Apr-2023